Aufbauend auf den ersten Teil möchte ich diesmal etwas näher darauf eingehen, was bei sogenannten „genomweiten DNA-Analysen“ in welchem Umfang getestet wird und was verbreitete Vorurteile und Missverständnisse sein können, wenn man von „Gentests“ und deren Anwendung in der Zucht spricht.
Seit Teil I sind gut 2 Jahre vergangen und zwischenzeitlich hat sich viel getan, bezogen auf die Analysen selbst, auf die Stimmung innerhalb der Züchterszene, aber auch im kleinen Rahmen meiner eigenen Zuchtstätte. Der C-Wurf, der bereits Jahre im Vorfeld und unabhängig von Genanalysen geplant war, ist nun bald 4 Monate alt und der erste Whippetwurf (und bisher auch der einzige Wurf generell, den ich finden konnte), der vollständig bei MyDogDNA analysiert wurde und dessen Analysen öffentlich zugänglich sind. Welche spannenden und erfreulichen Ergebnisse die Untersuchungen brachten, werde ich aber in einem anderen Beitrag detailliert darstellen. Die Absichten, die ich mit dieser Wurfplanung verfolgt habe, konnten jedenfalls nachweislich sehr gut erfüllt werden.
Ursprünglich dachte ich, drei Teile zum Thema würden ausreichen – aber die Reaktionen auf Teil I und andere Beiträge wie die Erklärung zum Breeder Tool und Genetic Health Index haben gezeigt, dass es noch sehr viel Erklärungsbedarf gibt. Und weil ich diese genetischen Profile so sinnvoll finde und für zukunftsweisend halte, mache ich mir erneut die Mühe, und entspinne für manche hoffentlich Stück für Stück ein bisschen von diesem Wirrwarr. Das Thema ist jedoch so breit gefächert und umfangreich, dass auch diesmal nur Häppchen serviert werden können.
Wer bereits nach den ersten Zeilen gar nichts versteht, dem sei die Einführung noch einmal dringend ans Herz gelegt.
Ich verwende als Beispiel die Analyse MyDogDNA von Genoscoper aus Finnland, da ich sie mit meinen Hunden durchgeführt habe und es einige Whippets in der öffentlichen Datenbank gibt, die ich als Beispiele nutzen darf. Auch Embark in den USA oder Feragen in Österreich bieten ähnliche DNA-Analysen an, jedoch unterscheiden sie sich in Details, deren Vor- und Nachteile jeder selbst für sich abwägen muss. Unter anderem hätte ich eben nicht die Möglichkeit, Profile einzelner Hunde für jeden zugänglich zu machen und zu erklären. Aber weder bekomme ich für Erwähnungen hier Rabatte, noch werde ich anderweitig gesponsert. Leider 😉
Ich möchte mit diesem Artikel also keinem einen direkten Vorzug geben, wer sich für DNA-Analysen interessiert, muss sich bitte bei den entsprechenden Anbietern schlau machen und das für ihn ideale Angebot auswählen.
Zugegeben, sich unter all den Angeboten zurechtzufinden, ist für Laien nicht ganz einfach. Es tut sich ja viel auf dem Markt, es ist fast eine Art Wettrüsten, und wir sind noch lange nicht am Ende der Möglichkeiten.
Tatsächlich gelang es durch das Engagement einiger Privatpersonen, in den vergangenen ein bis zwei Jahren alleine in Deutschland mehrere hundert Windhunde über MyDogDNA zu testen und deren öffentlich bereitgestellte Daten für populationsgenetische und andere Analysen zu nutzen (siehe die Zuchtberichte zum Whippet und auch zum Greyhound, aber auch intern für bspw. Rassemeetings des DWZRV).
War Lori noch die erste Whippethündin in Österreich, die dort getestet wurde, und ergab die Suche nach Deckrüden im Breeder Tool gerade ein gutes Dutzend Treffer, sind es nun (Stand heute) 81 Rüden. 96 Hündinnen sind als Zuchthündinnen eingetragen. Tendenz stark steigend.
Selbst in Zuchtverbänden wie dem DWZRV oder dem WWCS bekommt die Analyse MyDogDNA langsam Raum und man kann Rabatte nutzen oder einen Link zum Profil des Hundes in der Deckrüdenliste eintragen lassen.
Selbstverständlich ruft das auch Gegenstimmen unter Windhundzüchtern auf den Plan und im vergangenen Jahr sorgte auch ein Artikel im renommierten „Nature“ für Aufregung. Ich habe dazu bereits im Juli 2018 etwas geschrieben, möchte aber erneut darauf hinweisen, da er von Kritikern noch immer unreflektiert genutzt wird.
Der Titel des Artikels „Pet genomics medicine runs wild“ (in etwa zu übersetzen mit „Genomische Tiermedizin außer Rand und Band“) bezieht sich auf die tatsächlich wie Pilze aus dem Boden schießenden Angebote sogenannter „direct-to-consumer (DTC) companies“, die Gentests direkt an Hunde- und neuerdings auch Katzenbesitzer verkaufen.
Diese Kritik ist sehr wichtig und „The Greyhound Show“ hat dankenswerterweise kurz zusammengefasst, was das Problem an der aktuellen Situation ist bzw. wie bereits daran gearbeitet wird, um sie zu verbessern. Beispielsweise mit der „Initiative zur Harmonisierung von Genstests für Hunde“ (HGTD)
Wir selbst als Züchter können nur indirekt zu einer sinnvollen Lösung beitragen, indem wir Labore unterstützen, die gemeinsam (!) an der Qualitätssicherung dieser Tests arbeiten. Eines dieser Labore ist Genoscoper, Anbieter der von uns genutzten Analyse MyDogDNA.
Was aber auch getan werden kann bzw. getan wird, so lange die Forderung nach speziell ausgebildeten Beratern noch nicht umgesetzt wurde, ist selbst beratend tätig zu sein und nicht Zweifel und Missgunst zu schüren.
Hinter „The Greyhound Show“ stehen Menschen, die mit ihrer Expertise auf diesem Gebiet (forschend und lehrend z.B. am Lehrstuhl für Molekulare Tierzucht und Biotechnologie der LMU München) kompetent genug sind, um Fragen zu beantworten. Sie bieten u.a. die neue Züchterschulung des DWZRV an, ermöglichen Rabattaktionen durch Sammelbestellungen und beraten auch darüber hinaus unermüdlich Interessierte. Für Züchter und Deckrüdenhalter oder einfach nur interessierte Menschen, die ihre Hunde testen lassen, wurde die geheime Facebook-Gruppe „Windhunde bei MyDogDNA“ eingerichtet, in der Ergebnisse, Studien und offene Fragen besprochen werden können – in einem geschützten Rahmen, denn es sind nur Mitglieder erlaubt, deren Hunde nachweislich getestet wurden.
Natürlich sind auch Beiträge wie dieser hier wichtig und selbstverständlich stehe ich besonders für Whippetleute (und zunehmend für Silken Windsprites) immer ausführlich Rede und Antwort. Genoscoper selbst ist sehr hilfsbereit und versorgt jeden Interessierten zuverlässig und schnell mit wissenschaftlichen Publikationen und Informationen, man muss also keine Scheu haben, schließlich ist man dort (potentieller) Kunde.
Aber auch wenn Geld keine Rolle spielt, sollte Hilfe selbstverständlich sein. Angeblich ist Hundezucht ja ein Hobby, und da muss es doch möglich sein, dass man sich gegenseitig unterstützt. Wer allerdings nicht fragt, der wird auch keine Unterstützung bekommen, und dann liest man doch immer wieder die merkwürdigsten Dinge über diese Tests oder über Testergebnisse…
Wir stehen jedenfalls am Beginn einer interessanten und hilfreichen Entwicklung und nur zuzuschauen, oder gar einfach weiterzuwurschteln wie bisher, ist für viele Rassen ein Weg in die Perspektivlosigkeit.
Zu diesem Schluss kommen ja auch die Autoren des Artikels, die dabei nicht nur Tiere im Fokus haben (Übersetzung von mir): „Richtig durchgeführt, kann die Anwendung genetischer Tests bei Haustieren ein wirksamer Weg sein, Menschen besser mit den Möglichkeiten der Behandlung genetischer Erkrankungen vertraut zu machen. Falsch durchgeführt, könnte es das Vertrauen einer zunehmend wissenschaftsskeptischen Öffentlichkeit in die Wissenschaft untergraben.“
Was testen wir also? Oder besser, was testen wir nicht? Und was macht man mit den Ergebnissen?
Vielleicht ist es gut, kurz ein paar Worte zur Analyse an sich zu verlieren:
Mit Profil, Panel, Analyse, Test meine ich das:
Die Analyse von ausgewählten SNPs und von bekannten Mutationen, sei es im Bezug auf Farbe, Morphologie oder mit Erkrankungen verbundenen Mutationen.
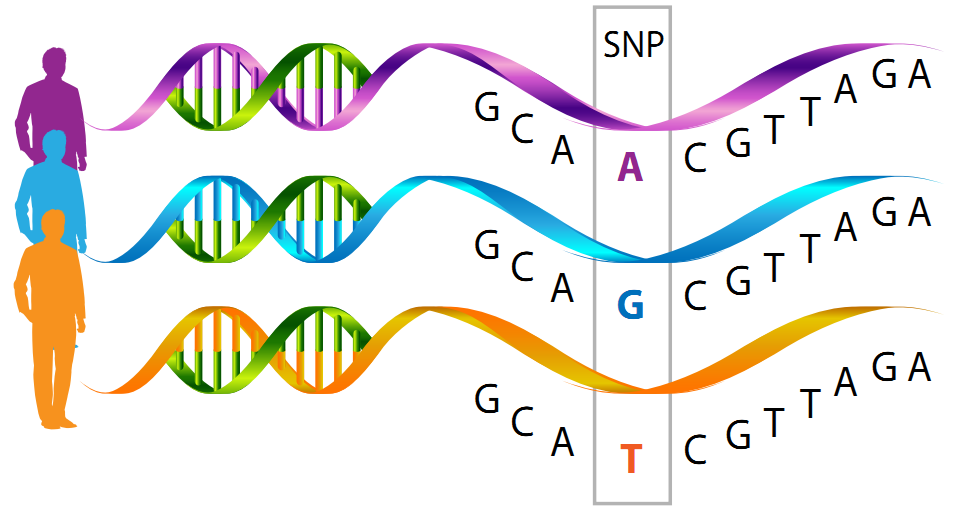
SNP, was ist das nun wieder?
SNP steht für Single Nucleotide Polymorphism (oder Einzelnukleotid Polymorphismus), wir nutzen im Sprachgebrauch einfach „Snips“, denn mit dem gesprochenen S N P im Wortfluss bricht sich eines Tages noch jemand die Zunge.
Im Text wird auch der Begriff Marker verwendet, was hier SNPs meint.
Ein Nukleotid ist also eine Struktureinheit der DNS (Desoxyribonukleinsäure, heute eigentlich nur mehr in englischer Form als DNA für acid = Säure bezeichnet) und besteht aus einem Zucker (eben der Desoxyribose), den vier Nukleinbasen A(denin), G(uanin), C(ytosin), T(hymin) und Phosphorsäure.
Diese Basen paaren sich immer miteinander – G paart sich mit C, A mit T – so entsteht die allseits bekannte Doppelhelix der DNA.
Ändert sich eine Base, ändert sich damit auch ihr Gegenpart.
Die Entdeckung dieser Struktur 1953 war, wie man sieht, bahnbrechend für die genetische Forschung und wurde damals übrigens ebenfalls im erwähnten „Nature“ publiziert, auf ganzen zwei Seiten 😉
Gene, bestimmte Abschnitte auf der DNA, codieren jedenfalls bekanntermaßen für Proteine, die den Körper formen und am Leben erhalten (s. Teil I).
Mit einer Änderung von Basenpaaren ändern sich womöglich Funktionen – oder eben auch gar nichts. Das hängt unter anderem davon ab, in welchem Bereich des Genoms Veränderungen stattfinden. SNPs innerhalb codierender Regionen können dazu führen, dass die gebildeten Proteine ein wenig anders aussehen und anders funktionieren, als bei anderen Lebewesen. Wir erinnern uns wieder, dass das ein Vorteil sein kann, aber auch ein Nachteil. Beim Menschen werden bestimmte SNPs mit einem erhöhten Risiko für entzündliche Darmerkrankungen, Diabetes Typ 1, Psoriasis oder andere Erkrankungen mit autoimmunem Hintergrund in Zusammenhang gebracht. Diese Formulierung ist besonders wichtig auch für Züchter, denn es gibt einen Unterschied zwischen einem erhöhten Risiko aufgrund genetischer Disposition und einer Mutation, die immer zu einer Erkrankung führt. Worauf wir Züchter unsere Hunde testen, sind meist solche monogenen (also nur durch ein Gen ausgelöste) Erkrankungen oder Eigenschaften, wie Farben, Felllänge und Struktur etc. Es gibt aber auch Erkrankungen wie Typen der PRA (Progressive Retinaatrophie) oder Cystinurie, bei denen erst die Analyse mehrerer beteiligter Gene/Mutationen Aufschluss bringt. Bei der PRA sind es aktuell über 20 bekannte Mutationen und beteiligte Gene.
Die Stärke der Ausprägung resp. die Schwere der Erkrankung hängt bei manchen Mutationen aber auch wieder von Begleitfaktoren ab, wie wir später am Beispiel des Faktor VII-Mangels sehen werden.
SNPs zeigen aber erst mal nur Variationen in einzelnen Basenpaaren und damit Unterschiede im Genom an, einzelne Nukleotide sind polymorph, also vielgestaltig.
Und diese Unterschiede sind für uns interessant.
Mit der Analyse MyDogDNA werden nicht nur eine ganze Reihe bekannter und problematischer Punktmutationen (an einer Nukleinbase), Indels (also Insertionen = Veränderungen durch zusätzliche Nukleotide oder Sequenzen und Deletionen = Löschung von Sequenzen, s. Teil I), Kopplungsmarker und andere Veränderungen der DNA untersucht, sondern auch über 20.000 standardisierte SNPs.
Besonderes Augenmerk wurde auch auf die DLA-Region, die für das Immunsystem zuständig ist, gelegt. Dazu und zum Rest aber in einem anderen Beitrag mehr.
Aus der Analyse der SNPs lassen sich Aussagen zum Inzuchtniveau/genomischen Inzuchtkoeffizient, zur genomweiten Diversität, zur Diversität innerhalb der Rasse und im Vergleich mit anderen Rassen und zur Verwandtschaften innerhalb der Rasse und zu anderen Rassen treffen.
Denn jede Rasse, die ja eine Subpopulation innerhalb der Haushunde darstellt, hat ihre eigenen populations- bzw. eben rassespezifischen Haplotypen, damit also Gruppen von Individuen, die die selbe Nukleotidsequenz auf einem Chromosom haben.
Ein Knackpunkt. Züchterisch wie allgemein für die Rassen.
Aus dem letzten Absatz ergibt sich auch, wie diese Analysen Einkreuzungen aufzeigen können, und zwar natürlich weit besser, als das der von vielen verspottete MARS-Test zur Rassereinheit mit nur 321 getesteten SNPs konnte. Im Vergleich noch einmal die Erinnerung: MyDogDNA testet aktuell über 20.000 SNPs.
Man könnte nun sagen, und das ist eine beliebte Kritik, 20.000 SNPs sind noch immer nicht genau genug, da muss schon eine komplette Sequenzierung des Genoms her! Oder, zumindest wie beim Menschen, irgendwas in der Größenordnung mehrerer 100.000 bis zu 1 Mio. SNPs.
Das ist jedoch nicht nötig, wie in diesem Artikel „Der Hund als Modell für den Humangenetiker“ ausführlich dargestellt wird. Ein kurzes Zitat:
Für einfach mendelnde Merkmale braucht man nur 10 bis 20 Hunde und einen Chip mit etwa 15.000 bis 30.000 SNPs, um das Merkmal völlig zweifelsfrei im Genom zu kartieren. Mit diesem ersten Schritt ist das Merkmal relativ grob im Genom des Hundes zugeordnet. Für eine exakte Feinkartierung untersucht man in einem zweiten Schritt Tiere von anderen Hunderassen, die zufällig auch dieses Merkmal zeigen. Das klingt zunächst überraschend, weil man glaubt, diese Mutation kommt nur bei der spezifischen Hunderasse vor. Aber dazu muss man wissen, dass nicht Neumutationen zu den heutigen Rassen geführt haben, sondern dass die selektive Züchtung von in der Spezies Hund bereits vorhandenen Varianten zur einheitlichen Rasse führte. Die sehr verschiedenen Hunderassen spiegeln die natürliche Variation in der Ausgangspopulation der Hunde wieder.
Forschung am Hund unterstützt nebenbei bemerkt die Forschung am Menschen und hilft damit tatsächlich nicht nur Hundezüchtern.
Aus den Daten, die wir über MyDogDNA zur Verfügung stellen, werden laufend wissenschaftliche Arbeiten erstellt, die peer-reviewed in Zeitschriften publiziert werden. Auf dem Blog von Genoscoper kann man die Veröffentlichungen und die Arbeiten an Projekten verfolgen.
Ein weiterer Kritikpunkt richtet sich gegen die Tests auf Defekte an sich, besser gesagt, auf die Zuchtplanung anhand von Gesundheitsergebnissen.
Jetzt mag man erst mal meinen, dass das an sich ja nicht schlecht wäre, Zucht nach Gesundheitsergebnissen.
Jein.
Zwar sind viele der bekannten Erkrankungen auf monogene, rezessive Defekte zurückzuführen (s.o.), die Genetikerin Carol Beuchart (Institute of Canine Biology) spricht sogar von etwa 70 bis 80% der Erkrankungen. Der Rest allerdings nicht – und vor allem kennen wir bisher nur einen Bruchteil davon.
Je mehr wir aufgrund von Gentests selektieren und womöglich symptomfreie Träger aus der Zucht ausschließen, desto schwieriger wird es, gesunde Hunde zu züchten. Denn diese Hunde tragen ja nicht nur dieses eine schadhafte Allel, sondern jede Menge wertvoller Gene, die damit verloren gehen.
Natürlich muss man überlegen, womit man es zu tun hat. Handelt es sich um eine Erkrankung wie die beim Whippet neu mit MyDogDNA entdeckte Bluterkrankheit Faktor VII-Mangel, die offenbar nur sehr milde Symptome auslöst und einen sehr geringen Krankheitswert hat, dazu auch nur von ganz wenigen Hunden getragen wird, oder hat man es mit etwas Ernsthaftem zu tun, wie bspw. der Greyhound Neuropathie (siehe Wie aus dem Nichts? Was wir aus Gendefekten lernen könnten)?
Als die Myostatin-Mutation, die für sogenannte „Bully-Whippets“ sorgte (siehe Myostatin-Mutation beim Whippet), vor einigen Jahren aufkam und ein Gentest dafür etabliert wurde, brach unter Whippetleuten ja teilweise rechte Panik aus. Blitzartig wurden Bestimmungen erlassen, alle Hunde in der Zucht und im Sport zu testen, um Träger auszuschließen. Gefunden hat man nur eine Handvoll Hunde (umgangssprachlich, genaue Zahlen sind zumindest mir nicht bekannt), der Verlust für die Rasse hielt sich also in Grenzen. Wirklich gut nachgedacht wurde aber eher nicht.
Der einzige Weg, weg von immer mehr Defekten zu kommen, ist die Zucht auf genetische Diversität.
Rein rechnerisch ist die Sache nämlich recht einfach: Einen großen Anteil der rezessiven, krankmachenden Mutationen kenne ich aktuell noch nicht, kann also nicht darauf testen. Sehen kann ich sie am Träger auch nicht. Züchte ich mit eng verwandten Hunden, bspw. mit einem COI von 25%, erben die Welpen mit einer Wahrscheinlichkeit von 25% zwei identische Kopien eines Allels, sind also homozygot in diesem Bereich. Sind es zufällig Allele, die geschädigt sind, bleibt dem Hund keine „gesunde“ Kopie mehr und eine Erkrankung prägt sich aus.
Gentests schützen also nur bedingt davor, kranke Hunde zu züchten, dennoch sollten sie auf jeden Fall durchgeführt werden, wenn sie rassespezifisch sind. Dazu gibt es auch einen aktuellen Beitrag: Fast 20 Jahre DNA-Tests – was können wir daraus lernen?
Ähnlich wie bei der Problematik um das unübersichtliche Angebot von Genanalysen muss man auch hier sagen, dass sich sehr viele Artikel von Wissenschaftlern und Laien genau auf dieses Thema richten, dass nämlich Tests auf monogene Defekte alleine keine Heilsbringer sind. Wer sich auch nur oberflächlich damit auseinandersetzt oder auseinandersetzen will und nicht mit Scheuklappen an das Thema herangeht, weiß das.
Der dritte große Kritikpunkt wäre die Validität: Sind Tests auf monogene Defekte überhaupt zuverlässig, wie ist es um die Qualität der Tests und der Wissenschaft dahinter bestellt?
Die meisten Defekte, die MyDogDNA testet (es sind aktuell über 150), sind nicht rassespezifisch. Rassespezifisch und nur beim Whippet vorkommend ist aktuell die erwähnte Myostatin-Mutation. Zu PFKD gibt es meines Wissens nur eine Fallstudie mit zwei Brüdern, gelistet wird sie von sämtlichen Laboren jedoch als rassespezifische Erkrankung.
Dennoch laufen alle Mutationen bei den Analysen mit, ganz egal, um welche Rasse oder welchen Rassemix es sich handelt, und das ist gut. Denn so hat man eben bspw. die Bluterkrankheit Faktor VII-Mangel beim Whippet durch eine MyDogDNA-Analyse neu entdeckt. Hunde, die zwei defekte Allele tragen, zeigen Blutgerinnungsstörungen und damit starke Blutungen nach Operationen oder schweren Verletzungen.
Diese Mutation kennt man von anderen Rassen und geht aktuell davon aus, dass es sich um eine recht alte Mutation handelt, die daher in vielen Rassen vertreten sein wird. Nun weiß man, der Whippet trägt sie auch, und er war bei weitem nicht die einzige Rasse, bei der es zur „Neuentdeckung“ durch MyDogDNA kam. Und Faktor VII-Mangel ist auch nicht die einzige Mutation, siehe die Arbeit „Genetic Panel Screening of Nearly 100 Mutations Reveals New Insights into the Breed Distribution of Risk Variants for Canine Hereditary Disorders„, die aus der Auswertung von Probennahmen zwischen 2013 und 2015 erfolgte. Der Whippet war da noch gar nicht dabei.
Interessanterweise scheint es so zu sein, dass der Effekt der Mutation beim Whippet tendenziell gering ist, während er bei anderen Rassen oft zu deutlich ausgeprägteren Symptomen führt. Wie ausgeprägt die Symptome im Einzelfall sind, lässt sich jedoch nicht vorhersagen und hängt vom Individuum selbst ab. Ob die Neigung zu postoperativen Blutungen bei Whippets auch daher rühren kann? Es würde eine Analyse erfordern.
Der Faktor VII-Mangel ist demnach ein Beispiel für eine Mutation, die von weiteren Faktoren beeinflusst wird, und nicht nur von dieser einzelnen Mutation.
Andere Mutationen, wie bspw. der als Medikamentenunverträglichkeit bekannte Defekt des MDR1-Gens (siehe MDR1-Defekt beim Windhund? Nein!), sind mutmaßlich noch älter und finden sich ebenfalls bei zahlreichen Rassen bzw. ganzen Rassegruppen. Gleiches gilt für die CEA, die Collie Eye Anomalie, deren Namen auf die betroffene Rassegruppe hinweist. Sie legen oftmals Zeugnis davon ab, dass bestimmte Rassen bei der Entstehungsgeschichte einer jüngeren Rasse beteiligt waren.
Bei MDR1 ist das im Bezug auf den Windhund konkret beim Silken Windsprite (ehemals Langhaarwhippet) der Fall, dessen Entstehungsgeschichte ja anders ablief, als vom Gründer der Rasse und seinen Anhängern behauptet. Diese Rasse ist nämlich keine langhaarige, aber dennoch reinrassige Varietät des Whippets, sondern entstand aus Whippets, die den Defekt nicht tragen, und Shelties, die diesen Defekt sehr häufig tragen. Vor der Möglichkeit, Hundegenome ganz einfach und kostengünstig analysieren zu lassen, war das Vorhandensein des MDR1-Defekts in der Population der indirekte Nachweis. Heute braucht man sich darüber gar nicht mehr zu streiten, die Analysen sind eindeutig und zeigen die (nur mehr weitläufige, aber dennoch vorhandene) Verwandtschaft zum Sheltie und Barsoi, und Probleme haben damit nur mehr ewig Gestrige.
Zu sehen sind im Vergleich auch hier wieder die Hunde (blaue Punkte sind Silken Windsprites), die sich auf halbem Wege zum Whippet (pink) befinden – es sind Hunde, die aus Einkreuzungen von Whippets zur Erhöhung der genetischen Diversität beim Silken Windsprite hervorgegangen sind. Interessant ist auch die Tabelle, wer sich genauer damit auseinandersetzen will, der kann das direkt unter diesem Link.

Wer sich konkret dafür interessiert, auf welcher Basis die Untersuchungen zu einzelnen Mutationen (Krankheiten, Medikamentenunverträglichkeiten) durchgeführt werden, welche Marker untersucht werden usw., wird in der öffentlichen Datenbank von MyDogDNA schnell fündig. Unter jeder Mutation findet sich ein Link zu einer pdf-Datei, in der das Merkmal in seinen Eigenschaften kurz beschrieben wird, betroffene Rassen genannt werden und es Verweise zu den herangezogenen Studien gibt.

Beispiele sind:
PFKD, beim Whippet in zwei Fällen beschrieben, aber dennoch rassespezifisch:
https://www.mydogdna.com/crm/disorders/128_PFKM_3breeds/en/128_PFKM_3breeds.pdf
Myostatin-Mutation aka Bully Whippet Syndrom, bisher exklusiv bei der Rasse Whippet aufgetreten:
https://www.mydogdna.com/crm/disorders/129_MSTN_new/en/129_MSTN_new.pdf
Faktor VII-Mangel:
https://www.mydogdna.com/crm/disorders/163_cFVII/en/163_cFVII.pdf
Wird, wie oben erwähnt, eine Mutation neu in einer Rasse entdeckt, kommt sie auf die „Watchlist“ und es werden tiefergehende Analysen vorgenommen. Daraus entstehen dann auch Arbeiten wie die weiter oben verlinkte, die ebenfalls wieder veröffentlicht werden. Für die Rasse selbst werden sie als „new potential disorders in the breed“, also potentiell neue rassespezifische Erkrankungen geführt. Stellt es sich heraus, dass die Mutation in der Rasse tatsächlich vorkommt und nicht auf kürzlich vorgenommene Einkreuzungen zurückzuführen ist, wird sie irgendwann den Status der „known disorders in the breed“, also bekannte Erkrankungen geführt.
Es wird außerdem ausgewiesen, und das gilt ebenfalls für die unter „Traits“ angeführten Farben, Felllänge, Größe etc., wie viele untersuchte Hunde der Rasse und allgemein in der Datenbank diese Mutation aufweisen. 2018 wurde auch die Arbeit „Frequency and distribution of 152 genetic disease variants in over 100,000 mixed breed and purebred dogs“ veröffentlicht, die auf die US-amerikanische Version MyBreedData zurückgreift.
Für den Dilute-Faktor, der zur Farbverdünnung und damit zu „blauen“ Whippets in allen Spielarten führt, liest man bspw.:

Der allerdings wirklich wichtige Aspekt bei der Analyse ist, und das möchte ich wieder einmal betonen, die ermittelte genetische Diversität und das Breeder Tool, das die analysierten Daten der Hunde miteinander abgleicht. Das ist etwas, was wir ohne DNA-Analysen nicht tun können und es zeigt sich immer wieder, und auch bei meinem C-Wurf, dass die mathematische Theorie hinter Inzuchtkoeffizient und Ahneverlust eben nur Anhaltspunkte liefert, aber nicht die Realität abbilden kann. Die Unterschiede zwischen Geschwistern sind teilweise erstaunlich und faszinierend sind die Vergleichsmöglichkeiten.
Siehe dazu auch noch einmal den Beitrag MyDogDNA, das Breeder Tool und der Genetic Health Index.
In Summe bieten sich also eine Fülle an Möglichkeiten, die gar nicht auf einen Schlag zu überblicken sind, und es gibt genauso zahlreiche Möglichkeiten, sich zu informieren und sich Hilfe zu holen, wenn es Fragen oder Verständnisprobleme gibt.
Ich finde, dass eine gute Portion Skepsis eine sehr wertvolle Eigenschaft darstellt und die sollte man sich immer behalten. Einfach neue Möglichkeiten in der Hundezucht ungenutzt an einem vorbeiziehen zu lassen, ist dagegen keine gute Idee. Vielleicht hat dieser Beitrag für manche ein wenig Hilfe bereitgestellt oder auch die Neugier geweckt, sich mit dem Thema auseinanderzusetzen und sich auf einen neuen Weg zu machen. Aufhalten kann man diese Entwicklung zumindest nicht mehr 🙂

Guten Tag, ich lese Ihre sehr interessanten und lehrreichen Artikel nun schon seit einiger Zeit. Die Veröffentlichung, die ich jetzt erhalten habe -Genomweite DNA-Analysen als Möglichkeiten in der modernen Hundezucht II hat einige Verweise auf frühere Veröffentlichungen, von denen ich leider keinen aufrufen kann. Auch bei der direkten Suche auf Ihrem Blog komme ich nicht weiter, die Artikel werden dort nicht angezeigt.
Was mache ich falsch? Wo kann ich diese Artikel finden.
Herzlichen Dank
Mit freundlichen GrüÃen
Silvia Fahnemann D-Rastatt
Liebe Frau Fahnemann,
die Links sind so gesetzt, dass sie sich in einem neuen Tab öffnen, so verliert man im eigentlichen Artikel nicht den Faden. Es ist möglich, dass Ihr Browser das nicht zulässt, weil eine entsprechende Einstellung vorgenommen wurde. Normalerweise sollte daher zumindest ein kleines Fenster (oder eine andere Benachrichtigung) erscheinen, in dem Sie gefragt werden, ob die Seite einen neuen Tab öffnen darf. Das muss dann von Ihnen zugelassen werden. Auch müsste es möglich sein, ein Häkchen zu setzen und für diesen Blog das Öffnen neuer Tabs generell zu erlauben.
Alternativ hilft ein Rechtsklick auf den gesetzten Link und die Auswahl „In neuem Tab/Fenster öffnen“.
Ich hoffe, das hilft und es freut mich, dass Sie hier mitlesen!
Liebe Grüße
Sabine
Hallo Sabine,
habe Eure interessantes BlogPost zum Thema Genetik in der Hundezucht auf Evitas Blog reBLOGGED https://blog4evita.com/2019/03/05/genetik-genomweite-dna-analysen-hundezucht/
Merci et beste Grüße bT!NA et Evita 🐾💕🐾
Danke dir, freut mich sehr 😊
Liebe Grüße
Sabine